-
荧光显微借助荧光分子标记可对样品中特定结构进行特异性成像,被广泛应用在生物与医药领域。尤其是近30年来超分辨显微技术[1-5]的出现,使得人们能够以更高的空间分辨率完成活细胞亚细胞水平、分子水平的成像,探索其在多种生理和病理过程中发挥的重要作用。利用超分辨显微,研究人员记录了活细胞中的细胞膜、线粒体膜、溶酶体膜等纳米尺度的膜动力学过程[6];研究细胞饥饿状态下内质网和线粒体的相互作用等[7]。然而,要在活细胞内实现染色体和基因位点的精准定位观测,尚需结合更高效无创的生物学手段。
CRISPR/Cas9 (clustered regularly interspaced short palindromic repeat/CRISPR-associated)系统是近年来出现的基因编辑技术。该系统由具有核酸酶活性的Cas9蛋白和具有序列靶向性的单链引导RNA (sgRNA)构成[8],sgRNA 决定了其特异性,能引导Cas9蛋白切割DNA双链。2012年,Jennifer Doudna和Emmanuelle Charpentier揭示了CRISPR/Cas9系统可以靶向切割外源DNA片段[9],随后张峰实验室首次将该系统应用于哺乳动物细胞内[10],进一步促使CRISPR技术在生物医学基因组工程方面的应用。同年Jinek团队改造研发出失去核酸酶活性但仍保留靶向目标DNA的定位功能的dCas9(dead Cas9)[11]蛋白,可以通过dCas9的精准定位功能实现对目标染色体和/或基因位点的特异性荧光标记,这拉开了CRISPR/dCas9系统的活细胞成像技术应用的序幕[12]。2013年,Chen等人运用dCas9的精准定位功能对靶基因位点进行荧光标记,实现了基于CRISPR/Cas9系统的活细胞成像[13]。
文中将对基于CRISPR/dCas9系统的活细胞成像技术进行分类介绍,阐述CRISPR/dCas9系统的活细胞成像技术的研究进展,并进一步总结该领域拟待解决的科学问题,以期为其进一步发展提供参考。
-
目前,应用于分子水平成像的技术主要有荧光原位杂交(fluorescent in situ hybridization,FISH)、LacO/LacI系统、TetO/TetR系统,以及基于基因编辑技术的锌指蛋白(zinc finger proteins,ZFP)、转录激活因子效应物(transcription activator-like effectors,TALEs)、CRISPR/Cas9系统等。FISH通过结合DNA探针实现对基因结构的成像,但此结合需通过甲醛或加热变性实现,可导致染色体结构的改变,不适用研究活细胞中的天然染色质动力学[14]。LacO/LacI系统和TetO/TetR系统通过在基因位点整合人工DNA序列阵列,使之与带有荧光分子标记的配体结合实现基因组成像,但这些插入的外源序列可能会干扰目标位点并产生干扰[15]。ZFP和TALEs系统都可以标记重复序列,如实现端粒的可视化[16-17],但是,ZFP系统的设计复杂耗时且细胞毒性大,TALEs存在体积大、递送困难的问题。
而CRISPR/Cas9系统可与多种荧光分子融合表达,操作较为简便,定位较为精准,对活细胞生理状态影响较小,在活细胞成像中应用更加灵活,既可有效可视化基因组中的非重复序列,又能实现染色体动力学的实时追踪成像。与其他成像技术相比,基于CRISPR/Cas9的活细胞成像技术具有独特的优势和局限性(见表1)。
表 1 基因组成像方法的优势和局限性
Table 1. Advantages and limitations of genome imaging methods
Imaging techniques Methods Advantages Limitations FISH DNA probe Widely applications; Simple preparation of probe Fixed cells; Potentially DNA damage LacO/LacI system et al. Synthetic DNA sequence Living cell imaging Potentially exogenous sequence interference ZFP Fused with fluorescent protein High specificity; Applied to repeated sequences Complex design; High cytotoxicity; Can’t applied to repeated sequences TALEs Fused with fluorescent protein High specificity; Low cytotoxicity; Applied to repeated sequences Difficulty in delivery; Can’t applied to non-repeated sequences CRISPR/Cas9 system Multiple labeling modes Simple design; Low cytotoxicity; Applicable to various sequences Risk of off-target -
CRISPR/Cas9系统主要利用荧光分子标记目标靶点,由短波长光激发荧光分子,产生光学跃迁发出波长更长的荧光实现基因可视化。荧光分子主要包括荧光蛋白(FPs)、有机荧光染料、新型纳米材料等[18]。
-
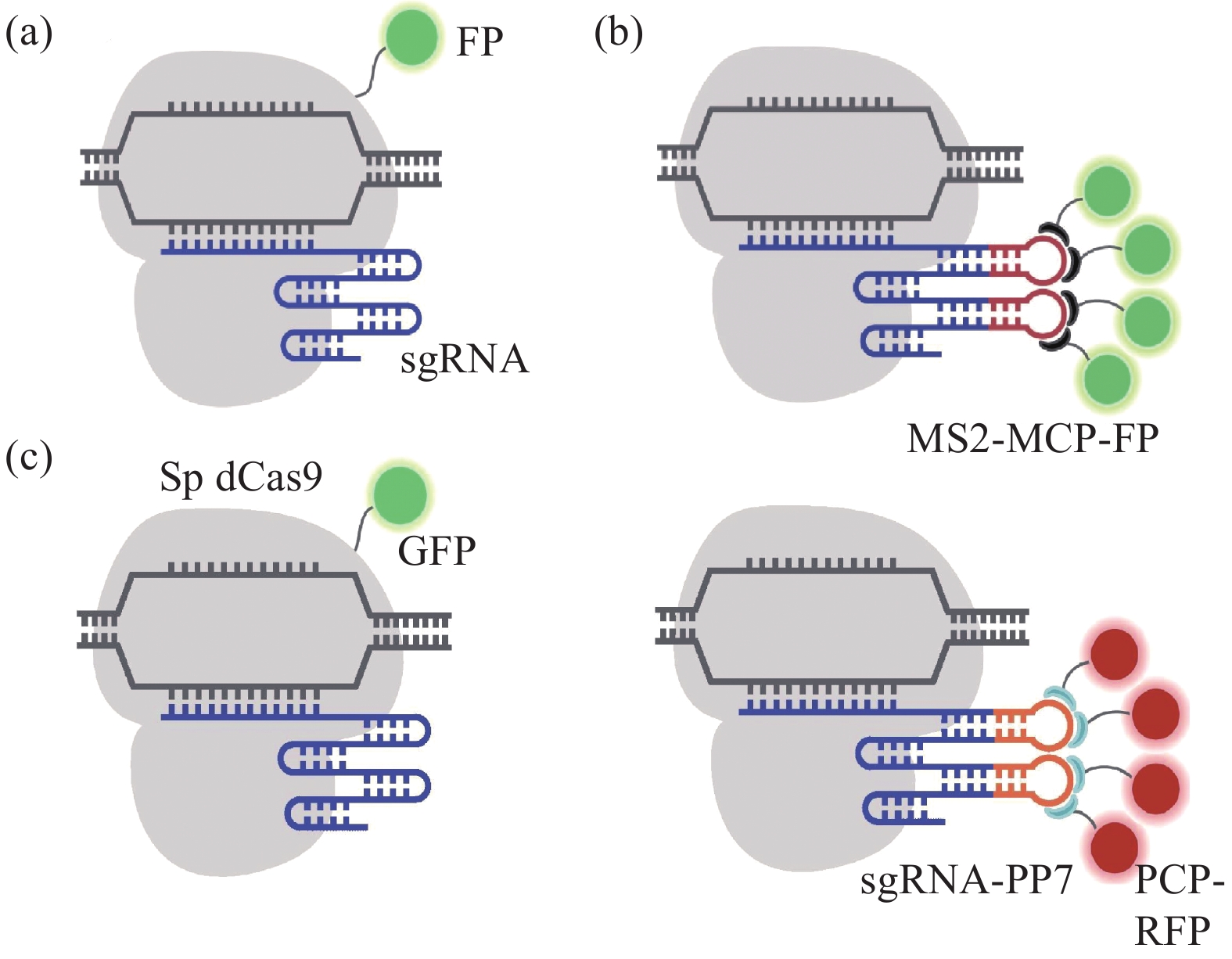
荧光蛋白具有本身生物毒性小、表达量高、荧光稳定、光漂白性弱、不影响生物功能等优点,在活细胞生物学功能的研究中应用最为广泛。通过将荧光蛋白与Cas9蛋白、sgRNA茎环支架耦联或同时修饰两者(图1(a)~(c)),完成对特定基因位点的标记应用于活细胞成像。并且该荧光耦联系统的逐步优化,使之不仅能富集荧光信号,降低荧光背景,提高信噪比,还可以实现多色成像,有助于分析染色体的三维结构。
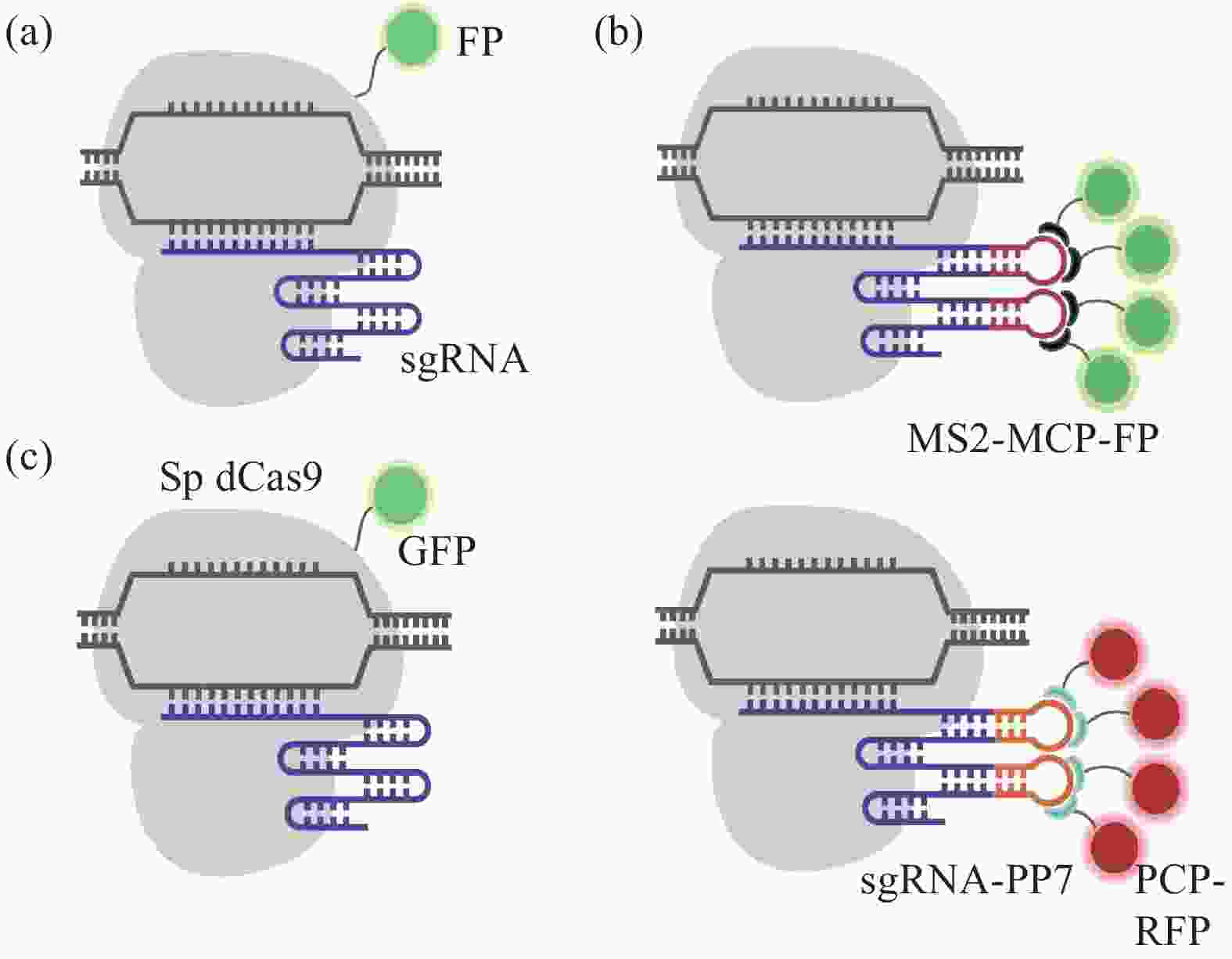
图 1 CRISPR/dCas9介导基于荧光蛋白的成像示意图[19]。(a) dCas9-FP融合蛋白; (b) MCP-MS2-FP系统:融合表达荧光蛋白的MCP适配体与被改造的sgRNA MS2茎环结合; (c) dCas9-GFP和PP7-PCP-RFP系统同时应用:多基因位点多色成像
Figure 1. Schematic diagram of CRISPR/dCas9 mediated fluorescent protein based on imaging[19]. (a) dCas9-FP fusion protein; (b) MCP-MS2-FP system: the MCP aptamer fused with fluorescent protein binds to the modified sgRNA MS2 stem ring; (c) Simultaneous application of dCas9-GFP and PP7-PCP-RFP systems: multicolor imaging of multiple gene loci
-
将Cas9直接与荧光蛋白如GFP、EGFP、mCherry融合形成Cas9-FP,并结合sgRNA实现对活细胞特定基因的动态成像。
2013年,Chen团队首次将缺乏核酸内切酶活性的dCas9蛋白与增强型绿色荧光蛋白(EGFP)融合,通过dCas9-EGFP与序列特异性sgRNAs的共表达对目标基因组进行成像,成功观察到细胞端粒的动力学变化、MUC4 基因座的亚核定位及其DNA复制时姐妹染色单体联会现象(图2(a)~(c))[13],这也是科学家首次利用CRISPR/Cas9技术实现了基因组的动态荧光标记,建立了第一个基于CRISPR/Cas系统的成像系统。随后,Ronald D. Vale团队于2014年构建了SunTag信号放大系统,该系统是由24个GCN4短肽和相应个数可特异性识别GCN4的单链可变区 (single-chain variable fragment, scFv)组成,通过将scFv-GCN4与荧光蛋白融合表达可同步实现定位和荧光信号放大两种功能,该团队将该SunTag系统与dCas9 融合(dCas9-SunTag),并在scFv-GCN4后面连接超级折叠绿色荧光蛋白(superfolder GFP, sfGFP)以标记端粒结构,与以往dCas9-EGFP直接融合的成像系统对比显著提升信噪比[20]。
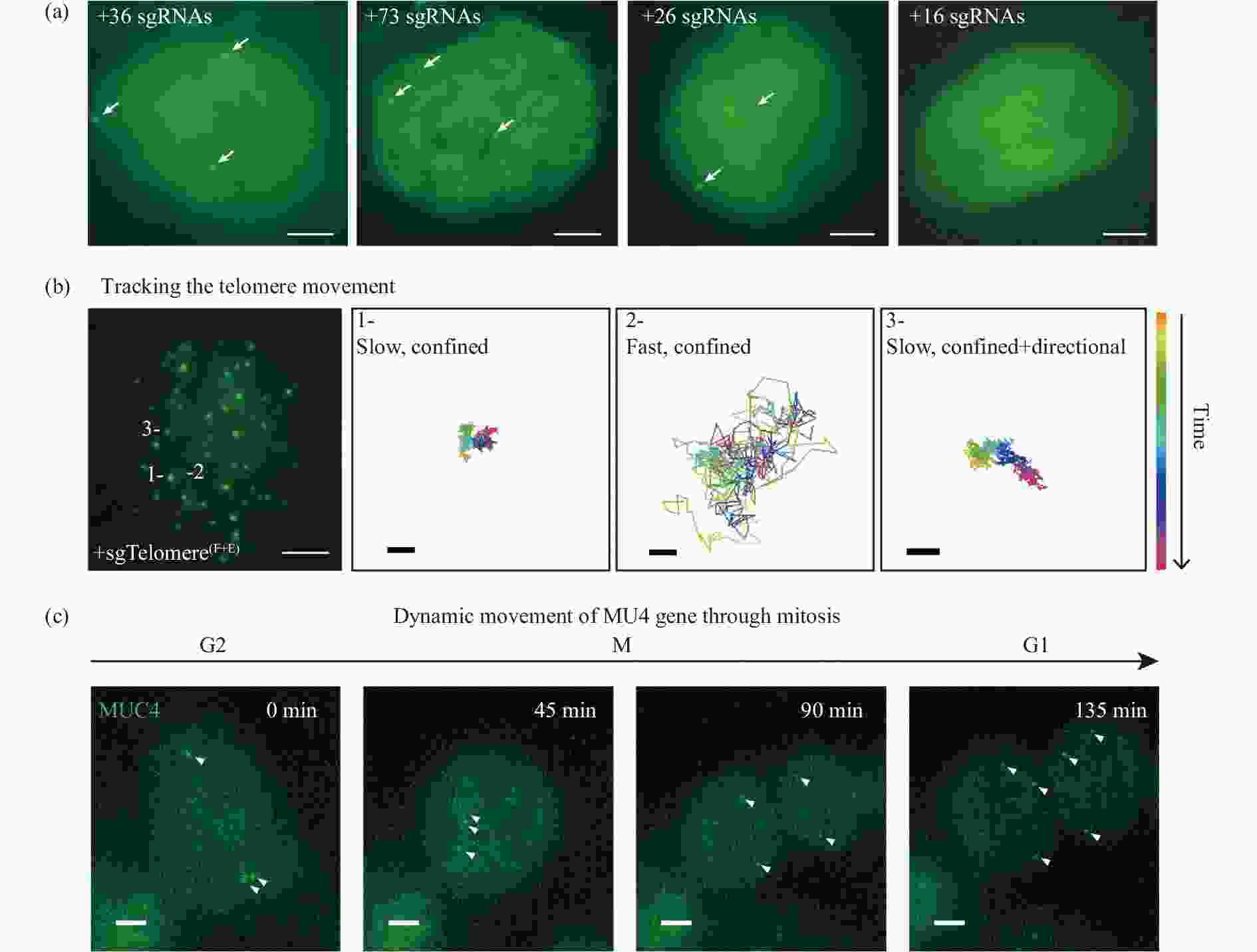
图 2 CRISPR/Cas9活细胞荧光成像图[13]。(a) 多个优化的sgRNAs对MUC4内含子的非重复区域进行CRISPR标记成像; (b) RPE细胞端粒的CRISPR成像(比例尺5 μm)和其运动轨迹示踪图(比例尺200 nm);(c) HeLa细胞有丝分裂时MUC4图像
Figure 2. CRISPR/Cas9 fluorescent imaging of living cells[13]. (a) CRISPR labeling of the nonrepetitive region of MUC4 intron using multiple optimized sgRNAs; (b) CRISPR imaging of telomeres in RPE cells (scale bar 5 μm) and its trajectory tracing diagram (scale bar 200 nm); (c) MUC4 image of HeLa cell during mitosis
由于sgRNA主导Cas9蛋白在基因组上的定位,因此它易于标记重复序列,单个sgRNA即可有效富集荧光信号。但对于可视化基因组中的非重复序列,往往需要共转染多个sgRNA,存在sgRNAs表达不同步,荧光强度弱的弊端。为了实现单个细胞内染色体非重复位点的高质量和高效率荧光标记,2018年,北京大学生物动态光学成像中心研究团队将多个sgRNA串联表达盒与dCas9-SunTag联合,规避了sgRNA表达不同步的问题,成功在活细胞中成像MUC4和HER2基因位点非重复序列[21]。
除此之外,为实现活细胞中RNA信号的动态检测,2020年,Sun等人将SunTag 系统与 CRISPR/Cas9 系统结合开发出一种新型RNA成像系统,并将其命名为CRISPR-Sunspot,即SunTag 介导的单分子RNA快照。该方法采用 24XGCN_v4 表位标签与 dCas9 的融合来招募 scFv-sfGFP 蛋白进行实现荧光信号的放大。 针对单个mRNA利用三个sgRNAs同时招募 dCas9-24 XGCN_v4-scFv-sfGFP 复合物,成功对U2OS细胞中的低丰度 mRNA进行成像[22]。
-
除修饰CRISPR/dCas9系统中Cas9蛋白外,还可以通过荧光蛋白修饰sgRNA实现活细胞成像。sgRNA由crRNA和tracrRNA (反式激活RNA)两部分组成,将MS2、PP7、boxB等插入tracrRNA具有的多个茎环结构中,使其可募集 RNA 结合蛋白MCP、PCP、λN22等,同时将RNA 结合蛋白与荧光蛋白融合实现基因位点的成像[23]。与基于dCas9成像系统进行比较,基于sgRNA成像系统在降低背景噪音的同时增强了荧光信号,具有更高的荧光恢复率,因此更适合于长时间的活细胞成像。
并且,这种成像方法可以利用多个适配体结合多个荧光蛋白,来达到增强荧光强度、降低背景信号目的。CRISPR介导的荧光原位杂交放大器CRISPR FISHer (CRISPR-mediated fluorescence in situ hybridization amplifier)就是利用dCas9联合带有两个PP7适配子修饰的gRNA(sgRNA-2 xPP7),通过招募PP7衣壳蛋白(PP7 coat protein, PCP)、GFP和T4 fibritin三聚体形成的融合蛋白foldon-GFP-PCP,从而实现单个sgRNA引导的非重复序列的稳健可视化,局部有效富集荧光信号,提高亮度和信号背景比(S/B ratio)[24]。
此外,还可以通过不同适配体与多种荧光蛋白的融合可以实现多重荧光成像,来实现分子间相互作用的追踪成像。Thoru Pederson等人开发了一种名为CRISPRainbow改进的多色CRISPR-dCas9 系统,用以对人U2OS 细胞中的基因组位点进行多重标记[25]。该系统利用RNA 适配体MS2、PP7或boxB来募集 MCP-BFP (蓝色荧光蛋白)、PPC-GFP (绿色荧光蛋白)或 kN22-RFP (红色荧光蛋白)以形成 RNA蛋白结合复合物。通过将MS2、PP7、boxB两两组合进一步将CRISPRainbow方法修改以生成双色标记系统。每个复合物募集其与荧光蛋白结合的相应蛋白质以形成MS2-MCP-BFP和PP7-PCP-GFP、PP7-PCP-GFP和boxB-kN22-RFP或boxB-kN22-RFP和MS2-MCP-BFP复合物,并且分别生成二次色青色、黄色或洋红色。而MS2-MCP-BFP、PP7-PCP-GFP和boxB-kN22-RFP与单个sgRNA的配对产生了三色标记系统,用于生成第三色,即白色。因此,CRISPRainbow 同时实现了对多达六个染色体位点进行成像,该系统也进一步丰富了基因组三维结构的荧光成像。
-
除单独修饰Cas9和sgRNA外,还可通过同时修饰Cas9和sgRNA实现多色标记[26]。活细胞中多种基因元素的同时追踪对于全面理解基因组结构的动态调控至关重要。Guan等人研究发现dCas9/sgRNA的成像系统可用于测量dCas9-sgRNA复合体在活细胞中的核动力学以及停留时间等[27]。
-
除以上介绍的融合荧光蛋白成像方法外,还可通过将有机染料与CRISPR/Cas9联合实现基因组标记。与荧光蛋白相比,有机染料尺寸小不易影响染色体的结构和功能,光毒性小、光稳定性强、量子产率高[28]。现已开发多种用于CRISPR/dCas9成像系统的有机染料,包括如基于Halo标签的系统、基于RNA适配子的系统和基于分子信标(MB)的系统。
HaloTag 是一种大小为33kDa肽标签,由细菌酶卤代烷脱卤酶和用于荧光发射的配体分子组成。该系统需未标记的sgRNA和 HaloTag 融合到 dCas9 的 C 端生成的 dCas9-HaloTag 融合蛋白,并通过有机荧光染料(Halo 配体)与 dCas9-HaloTag 的共价结合来发射荧光(图3(a))。Knight C.S.团队通过HaloTag示踪Cas9并成功观测到其运动轨迹及进入小鼠 3 T3 细胞后与染色质的结合效率[29]。基于RNA适配子的系统使用3,5-二氟-4-羟基亚苄基咪唑烷酮(DFHBI)染料(图3(b)),这种染料在生理条件下很容易猝灭,但当与其同源RNA适配子结合时会发光[29-30]。目前,基于Halo标签和基于DFHBI的CRISPR的标记系统都已被用于测量dCas9-sgRNA复合体在活细胞中的核动力学和靶上停留时间,揭示了CRISPR系统区分互补和不匹配目标的特征。但在基于Halo标签的系统中,由于未结合的荧光配体是非猝灭的,因此需要大量清洗才能从细胞中移除多余的配体,这可能会改变细胞生理并限制对染色质动力学的准确评估[31]。
图 3 CRISPR/dCas9介导基于有机染料的成像示意图[19]。(a) dCas9-HaloTag融合蛋白; (b) sgRNA-Broccoli/DFHBI-1T系统:经修饰的sgRNA用于结合一到多个RNA适配体DFHBI-1T;(c) sg-RNAMTS-MB系统:分子靶标MB与核酸靶点MTS结合激发荧光
Figure 3. Schematic diagram of CRISPR/dCas9 mediated organic dye based imaging[19]. (a) dCas9-HaloTag fusion protein; (b) sgRNA-Bro-ccoli/DFHBI-1T system: modified sgRNA is used to bind one or more RNA aptamers DFHBI-1T; (c) sgRNA-MTS-MB system: molecular biomarker MB combines with nucleic acid target MTS to stimulate fluorescence
而分子靶标(molecular biomarker, MB)是一种可猝灭的荧光寡核苷酸探针,当与互补核酸靶标结合时会激活荧光[19]。CRISPR/MB由dCas9、MB和含有唯一MB靶序列(MTS)的sgRNA组成(图3(c)),可对目标基因有更准确的定位,提高了对端粒内重复元件的时间分辨率[32]。RNA引导的核酸内切酶原位标记(RGEN-ISL),保留了染色质的自然结构,并通过应用两部分引导RNA和重组Cas9内切酶的复合体,允许特异性和同时原位检测多色基因组序列[33]。
-
量子点(quantum dots,QDs)是尺寸为1~20 nm的发光半导体纳米颗粒,因具有优于合成染料和荧光蛋白的亮度和光稳定性,使其成为需要敏感测量应用中的优秀探测器,被应用于体外单分子成像等[34]。2012年,量子点已被用于活细胞成像,研究者通过将 PrPc-DNA适配子于荧光量子点连接,用于追踪细胞内成像的动力学;在免疫荧光分析中,该复合体可共定位于溶酶体、内质网和高尔基体[35]。利用纳米粒子与适配子的耦联作用,在没有任何荧光标记的情况下,通过光学显微镜直接观察适配子的亚细胞定位[36]。CRISPR/Cas9介导量子点活细胞成像是通过基于硫辛酸连接酶(LplA)或基于生物素/链霉亲和素的方法在活细胞核中与dCas9结合实现的。一方面,硫辛酸连接酶可将与硫辛酸连接酶受体多肽融合的dCas9连接到反式环辛烯(TCO2)上,并将四嗪修饰的量子点导入细胞,通过Diels-Alder环加成反应来标记dCas9(图4(a))。另一方面,dCas9可融合到一个15个氨基酸生物素受体多肽(BAP)标签,然后在细胞中的生物素连接酶存在下进行生物素化[17],随后利用链霉亲和素修饰的量子点来标记dCas9蛋白(图4(b))。研究显示量子点标记CRISPR/dCas9双色成像系统可有效显示宿主细胞中的HIV前病毒DNA[17, 36]。此外,CRISPR-QD系统已被用于使用生物素化重组dCas9 (rdCas9-Bio)和链霉亲和素结合量子点(SA-Qds)实时监测伪狂犬病病毒(PRV)核酸[37]。量子点具有良好的光学特性,因此CRISPR/Cas9介导量子点的活细胞成像技术可减少对生理状态下活细胞基因组功能和活性的干扰,是单基因位点成像的有前途的候选方法。但由于量子点进入细胞后容易进入溶酶体以及易形成发光聚集体等缺点限制了其在基于CRISPR/Cas9的活细胞成像技术的应用[37]。
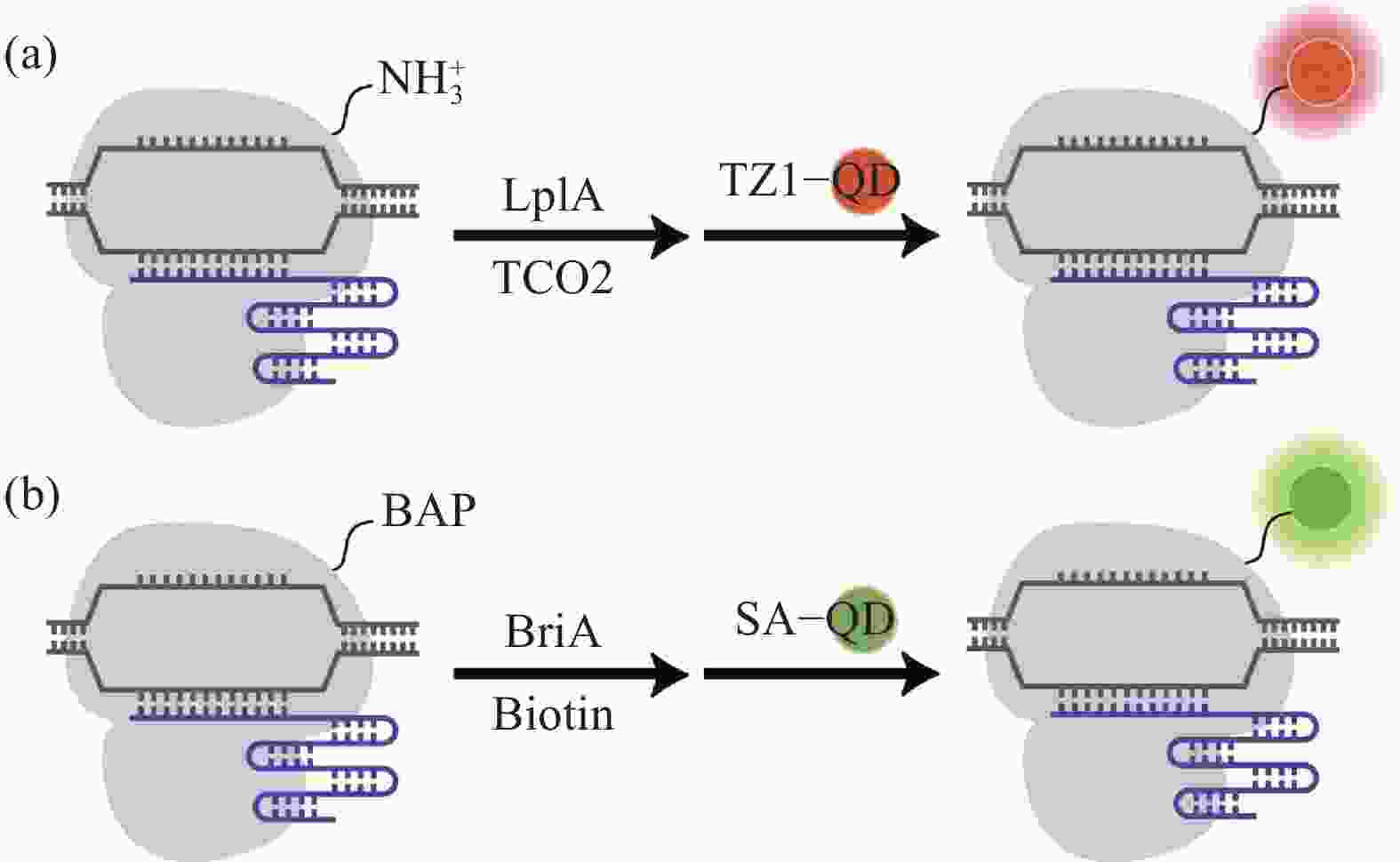
图 4 CRISPR/dCas9介导基于量子点(QDs)的成像示意图[19]。 (a) LplA介导量子点系统:通过LplA介导dCas9与TCO2结合并与TZ1-QD反应; (b) BirA介导量子点系统:同过BirA介导dCas9生物素化并与SA-QD结合
Figure 4. Schematic diagram of CRISPR/dCas9 mediated quantum dots (QDs) based imaging[19]. (a) LplA-mediated quantum dots system: dCas9 combines with TCO2 and reacts with TZ1-QD via LplA; (b) BirA-mediated quantum dots system: BirA-mediated dCas9 biotinylated and combines with SA-QD
-
可视化生物大分子以及观测它们在不同环境下相互作用的变化对深入探索生命奥秘具有不可替代的研究意义,由此,用以帮助理解细胞内染色质动力学变化、实时转录及翻译等不同生物学过程的清晰精准客观的超微活细胞成像,是研究人员一直追求的方向。活细胞成像技术已成为细胞生物学、遗传学、医学领域的必不可少的研究工具。
CRISPR/Cas9是具有强大编辑能力、使用范围广泛的基因编辑技术,现已被开发成为高效的活细胞成像工具,通过联合超分辨显微技术可成功实现对细胞染色质、基因组位点及RNA的精确可视化。现已开发多种基于CRISPR/Cas9系统动态观测染色体动力学和基因组时空变化的活细胞成像技术。此外,研究人员还利用不同来源的Cas9实现同时对单个/多个基因组位点进行双色或多色成像,并利用SunTag系统和针对sgRNA的修饰实现荧光成像效果的增强。
尽管作为现在细胞生物学研究的主要表征工具之一的超分辨荧光成像技术已取得重大进展,但仍需在荧光分子生物相容性、抗光漂白、荧光量子产率等方面做出突破。首先,荧光分子可能会影响生物的正常功能,其次,激发荧光产生的高光子通量会带来光毒性损害细胞,以及长时程采集荧光图像不可避免地使光漂白性能影响成像质量。再次,进一步扩大基于CRISPR/Cas9系统的新型活细胞成像技术的特异性和实用性是咎不可待的。例如,由于CRISPR/Cas9系统潜在的脱靶效应会导致细胞突变并产生不良影响[38]。该脱靶效应在成像时会增加假阳性位点,而降低成像的信噪比[39],因此未来仍需进一步加强对Cas9及sgRNA的有效改造以提高成像系统的特异性和信号背景比[40]。此外,由于CRISPR/Cas9系统最主要的递送形式为质粒和病毒载体[41],较低的递送效率以及外源DNA给机体带来潜在危害都限制了该技术的应用,开发递送效率更高、生物相容性更强、低毒性更低的CRISPR/Cas9活细胞成像系势在必行。
基于CRISPR/Cas9系统的活细胞成像技术,已用于帮助人们了解基因组中特异性位点的核定位及染色体时空的变化规律,但开发高透膜性、光稳定性强、荧光亮度高的荧光分子,提升CRISPR系统分子定位的精确度、降低脱靶率,探索安全有效的细胞内递送方式,对于实现活细胞长时程成像是重要且必要的。
Advances in live cell imaging technology of CRISPR/Cas9 system (invited)
-
摘要: CRISPR/Cas9系统因其高效、操作简便、物种适应性广等优势被广泛应用于基因编辑领域,该系统是由靶向目标DNA序列的引导RNA (sgRNA)和具有切割酶活性的Cas9蛋白组成。近年来,通过将核酸酶失活的Cas9突变体dCas9 (dead Cas9)或sgRNA与荧光蛋白(FPs)、有机染料、量子点(QDs)结合开发出一系列超分辨活细胞成像技术,该技术有助于在更高分辨率下研究不同基因、染色体以及基因与染色体之间的时空关系,对促进遗传学、细胞生物学和生物医学等领域的快速发展具有重要意义。文中主要总结基于CRISPR/Cas9系统的活细胞成像技术的最新进展,有望进一步扩大活细胞成像技术在生物医学领域的广泛应用。
-
关键词:
- 活细胞成像 /
- CRISPR/Cas9 /
- 荧光分子 /
- 超分辨
Abstract: CRISPR/Cas9 system are widely used in gene editing due to its high efficiency, simple operation and wide species adaptability. The system consists of a guide RNA (sgRNA) that targets the target DNA series and Cas9 with cleavage enzyme activity. In recent years, researchers have developed a range of super-resolution live cell imaging techniques by combining nuclease-inactivated Cas9 mutants dCas9 (dead Cas9) or sgRNA with fluorescent proteins (FPs), organic dyes, and quantum dots (QDs). This technology helps researchers to study different genes, chromosomes and the spatio-temporal relationship between genes and chromosomes at higher resolutions, which is of great significance to promote the rapid development of genetics, cell biology and biomedicine. This paper summarizes the advances in live cell imaging technology based on CRISPR/Cas9 system, which is expected to further expand the wide application in the biomedical field.-
Key words:
- live cell imaging /
- CRISPR/Cas9 /
- fluorescent molecules /
- super resolution
-
图 1 CRISPR/dCas9介导基于荧光蛋白的成像示意图[19]。(a) dCas9-FP融合蛋白; (b) MCP-MS2-FP系统:融合表达荧光蛋白的MCP适配体与被改造的sgRNA MS2茎环结合; (c) dCas9-GFP和PP7-PCP-RFP系统同时应用:多基因位点多色成像
Figure 1. Schematic diagram of CRISPR/dCas9 mediated fluorescent protein based on imaging[19]. (a) dCas9-FP fusion protein; (b) MCP-MS2-FP system: the MCP aptamer fused with fluorescent protein binds to the modified sgRNA MS2 stem ring; (c) Simultaneous application of dCas9-GFP and PP7-PCP-RFP systems: multicolor imaging of multiple gene loci
图 2 CRISPR/Cas9活细胞荧光成像图[13]。(a) 多个优化的sgRNAs对MUC4内含子的非重复区域进行CRISPR标记成像; (b) RPE细胞端粒的CRISPR成像(比例尺5 μm)和其运动轨迹示踪图(比例尺200 nm);(c) HeLa细胞有丝分裂时MUC4图像
Figure 2. CRISPR/Cas9 fluorescent imaging of living cells[13]. (a) CRISPR labeling of the nonrepetitive region of MUC4 intron using multiple optimized sgRNAs; (b) CRISPR imaging of telomeres in RPE cells (scale bar 5 μm) and its trajectory tracing diagram (scale bar 200 nm); (c) MUC4 image of HeLa cell during mitosis
图 3 CRISPR/dCas9介导基于有机染料的成像示意图[19]。(a) dCas9-HaloTag融合蛋白; (b) sgRNA-Broccoli/DFHBI-1T系统:经修饰的sgRNA用于结合一到多个RNA适配体DFHBI-1T;(c) sg-RNAMTS-MB系统:分子靶标MB与核酸靶点MTS结合激发荧光
Figure 3. Schematic diagram of CRISPR/dCas9 mediated organic dye based imaging[19]. (a) dCas9-HaloTag fusion protein; (b) sgRNA-Bro-ccoli/DFHBI-1T system: modified sgRNA is used to bind one or more RNA aptamers DFHBI-1T; (c) sgRNA-MTS-MB system: molecular biomarker MB combines with nucleic acid target MTS to stimulate fluorescence
图 4 CRISPR/dCas9介导基于量子点(QDs)的成像示意图[19]。 (a) LplA介导量子点系统:通过LplA介导dCas9与TCO2结合并与TZ1-QD反应; (b) BirA介导量子点系统:同过BirA介导dCas9生物素化并与SA-QD结合
Figure 4. Schematic diagram of CRISPR/dCas9 mediated quantum dots (QDs) based imaging[19]. (a) LplA-mediated quantum dots system: dCas9 combines with TCO2 and reacts with TZ1-QD via LplA; (b) BirA-mediated quantum dots system: BirA-mediated dCas9 biotinylated and combines with SA-QD
表 1 基因组成像方法的优势和局限性
Table 1. Advantages and limitations of genome imaging methods
Imaging techniques Methods Advantages Limitations FISH DNA probe Widely applications; Simple preparation of probe Fixed cells; Potentially DNA damage LacO/LacI system et al. Synthetic DNA sequence Living cell imaging Potentially exogenous sequence interference ZFP Fused with fluorescent protein High specificity; Applied to repeated sequences Complex design; High cytotoxicity; Can’t applied to repeated sequences TALEs Fused with fluorescent protein High specificity; Low cytotoxicity; Applied to repeated sequences Difficulty in delivery; Can’t applied to non-repeated sequences CRISPR/Cas9 system Multiple labeling modes Simple design; Low cytotoxicity; Applicable to various sequences Risk of off-target -
[1] Abbe E. Beiträge zur theorie des mikroskops und der mikroskopischen wahrnehmung [J]. Archiv für Mikroskopische Anatomie, 1873, 9(1): 413-468. [2] Huang B, Wang W, Bates M, et al. Three-dimensional super-resolution imaging by stochastic optical reconstruction microscopy [J]. Science, 2008, 319(5864): 810-813. doi: 10.1126/science.1153529 [3] Wei Tongda, Zhang Yunhai, Yang Haomin. Super resolution imaging technology of stimulated emission depletion [J]. Infrared and Laser Engineering, 2016, 45(6): 0624001. (in Chinese) doi: 10.3788/IRLA201645.0624001 [4] Yang Jianyu, Pan Leiting, Hu Fen, et al. Stochastic Optical Reconstruction Microscopy and its application [J]. Infrared and Laser Engineering, 2017, 46(11): 1103008. (in Chinese) doi: 10.3788/IRLA201746.1103008 [5] Zhang Zhimin, Kuang Cuifang, Wang Ziang, et al. Dual-color fluorescence emission difference super-resolution microscopy [J]. Chinese Optics, 2018, 11(3): 329-336. (in Chinese) doi: 10.3788/CO.20181103.0329 [6] Shim S H, Xia C, Zhong G, et al. Super-resolution fluorescence imaging of organelles in live cells with photoswitchable membrane probes [J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109: 13978-13983. doi: 10.1073/pnas.1201882109 [7] Zhu F, Yang Z, Wang F, et al. 4-Dimensional observation ER-mitochondria interaction in living cells under nanoscopy by a stable pyridium salt as biosensor [J]. Sensors and Actuators B:Chemical, 2020, 305: 127492. doi: 10.1016/j.snb.2019.127492 [8] Chylinski K, Makarova K S, Charpentier E, et al. Classification and evolution of type II CRISPR-Cas systems [J]. Nucleic Acids Research, 2014, 42(10): 6091-6105. doi: 10.1093/nar/gku241 [9] Pennisi E. The CRISPR craze [J]. Science, 2013, 341(6148): 833-836. doi: 10.1126/science.341.6148.833 [10] Shalem O, Sanjana N E, Hartenian E, et al. Genome-scale CRISPR-Cas9 knockout screening in human cells [J]. Science, 2014, 343(6166): 84-87. doi: 10.1126/science.1247005 [11] Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity [J]. Science, 2012, 337(6096): 816-821. doi: 10.1126/science.1225829 [12] Qiao H, Wu J, Zhang X, et al. The advance of CRISPR-Cas9-Based and NIR/CRISPR-Cas9-Based imaging system [J]. Frontiers in Chemistry, 2021, 9: 786354. doi: 10.3389/fchem.2021.786354 [13] Chen B, Gilbert L A, Cimini B A, et al. Dynamic imaging of genomic loci in living human cells by an optimized CRISPR/Cas system [J]. Cell, 2013, 155(7): 1479-1491. doi: 10.1016/j.cell.2013.12.001 [14] Beliveau B J, Boettiger A N, Avendaño M S, et al. Single-molecule super-resolution imaging of chromosomes and in situ haplotype visualization using Oligopaint FISH probes [J]. Nature Communications, 2015, 6: 7147. doi: 10.1038/ncomms8147 [15] Marklund E, Van Oosten B, Mao G, et al. DNA surface exploration and operator bypassing during target search [J]. Nature, 2020, 583(7818): 858-861. doi: 10.1038/s41586-020-2413-7 [16] Liu Y, Zhao N, Kanemaki M T, et al. Visualizing looping of two endogenous genomic loci using synthetic zinc-finger proteins with anti-FLAG and anti-HA frankenbodies in living cells [J]. Genes to Cells: Devoted to Molecular & Cellular Mechanisms, 2021, 26(11): 905-926. doi: 10.1111/gtc.12893 [17] Hu H, Zhang H, Wang S, et al. Live visualization of genomic loci with BiFC-TALE [J]. Scientific Reports, 2017, 7: 40192. doi: 10.1038/srep40192 [18] Liu Zhihe, Wu Changfeng. Advances in application of materials of super-resolution imaging fluorescent probe [J]. Chinese Optics, 2018, 11(3): 344-362. (in Chinese) doi: 10.3788/CO.20181103.0344 [19] Wu X, Mao S, Ying Y, et al. Progress and challenges for live-cell imaging of genomic loci using CRISPR-based platforms [J]. Genomics, Proteomics & Bioinformatics, 2019, 17(2): 119-128. doi: 10.1016/j.gpb.2018.10.001 [20] Tanenbaum M E, Gilbert L A, Qi L S, et al. A protein-tagging system for signal amplification in gene expression and fluorescence imaging [J]. Cell, 2014, 159(3): 635-646. doi: 10.1016/j.cell.2014.09.039 [21] Shao S, Chang L, Sun Y, et al. Multiplexed sgRNA expression allows versatile single nonrepetitive DNA labeling and endogenous gene regulation [J]. ACS Synthetic Biology, 2018, 7(1): 176-186. doi: 10.1021/acssynbio.7b00268 [22] Sun N H, Chen D Y, Ye L P, et al. CRISPR-sunspot: Imaging of endogenous low-abundance RNA at the single-molecule level in live cells [J]. Theranostics, 2020, 10(24): 10993-11012. doi: 10.7150/thno.43094 [23] Le P, Ahmed N, Yeo G W. Illuminating RNA biology through imaging [J]. Nature Cell Biology, 2022, 24(6): 815-824. doi: 10.1038/s41556-022-00933-9 [24] Lyu X Y, Deng Y, Huang X Y, et al. CRISPR FISHer enables high-sensitivity imaging of nonrepetitive DNA in living cells through phase separation-mediated signal amplification [J/OL]. Cell Research, (2022-09-14)[2022-10-10]. https://www.nature.com/articles/s41422-022-00712-z. [25] Ma H, Tu L C, Naseri A, et al. Multiplexed labeling of genomic loci with dCas9 and engineered sgRNAs using CRISPRainbow [J]. Nature Biotechnology, 2016, 34(5): 528-530. doi: 10.1038/nbt.3526 [26] Deng W, Shi X, Tjian R, et al. CASFISH: CRISPR/Cas9-mediated in situ labeling of genomic loci in fixed cells [J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(38): 11870-11875. doi: 10.1073/pnas.1515692112 [27] Guan J, Liu H, Shi X, et al. Tracking multiple genomic elements using correlative CRISPR imaging and sequential DNA FISH [J]. Biophysical Journal, 2017, 112(6): 1077-1084. doi: 10.1016/j.bpj.2017.01.032 [28] Wagh S B, Maslivetc V A, La Clair J J, et al. Lessons in organic fluorescent probe discovery [J]. Chembiochem, 2021, 22(22): 3109-3139. doi: 10.1002/cbic.202100171 [29] Knight S C, Xie L, Deng W, et al. Dynamics of CRISPR-Cas9 genome interrogation in living cells [J]. Science, 2015, 350(6262): 823-826. doi: 10.1126/science.aac6572 [30] Ma H, Tu L C, Naseri A, et al. CRISPR-Cas9 nuclear dynamics and target recognition in living cells [J]. The Journal of Cell Biology, 2016, 214(5): 529-537. doi: 10.1083/jcb.201604115 [31] Liu Y, Miao K, Dunham N P, et al. The cation-π interaction enables a Halo-Tag fluorogenic probe for fast no-wash live cell imaging and gel-free protein quantification [J]. Biochemistry, 2017, 56(11): 1585-1595. doi: 10.1021/acs.biochem.7b00056 [32] Mao S, Ying Y, Wu X, et al. CRISPR/dual-FRET molecular beacon for sensitive live-cell imaging of non-repetitive genomic loci [J]. Nucleic Acids Research, 2019, 47(20): e131. doi: 10.1093/nar/gkz752 [33] Ishii T, Schubert V, Khosravi S, et al. RNA-guided endonuclease-in situ labelling (RGEN-ISL): a fast CRISPR/Cas9-based method to label genomic sequences in various species [J]. The New Phytologist, 2019, 222(3): 1652-1661. doi: 10.1111/nph.15720 [34] Kumar Y R, Deshmukh K, Sadasivuni K K, et al. Graphene quantum dot based materials for sensing, bio-imaging and energy storage applications: a review [J]. RSC Advances, 2020, 10(40): 23861-23898. doi: 10.1039/d0ra03938a [35] Chen L Q, Xiao S J, Hu P P, et al. Aptamer-mediated nanoparticle-based protein labeling platform for intracellular imaging and tracking endocytosis dynamics [J]. Analytical Chemistry, 2012, 84(7): 3099-3110. doi: 10.1021/ac202810b [36] Ma Y, Wang M, Li W, et al. Live visualization of HIV-1 proviral DNA using a Dual-Color-Labeled CRISPR system [J]. Analytical Chemistry, 2017, 89(23): 12896-12901. doi: 10.1021/acs.analchem.7b03584 [37] Yang Y B, Tang Y D, Hu Y, et al. Single virus tracking with quantum dots packaged into enveloped viruses using CRISPR [J]. Nano Letters, 2020, 20(2): 1417-1427. doi: 10.1021/acs.nanolett.9b05103 [38] Asmamaw M, Zawdie B. Mechanism and applications of CRISPR/Cas-9-Mediated genome editing [J]. Biologics:Targets & Therapy, 2021, 15: 353-361. doi: 10.2147/BTT.S326422 [39] Nelles D A, Fang M Y, O'connell M R, et al. Programmable RNA tracking in live cells with CRISPR/Cas9 [J]. Cell, 2016, 165(2): 488-496. doi: 10.1016/j.cell.2016.02.054 [40] Lazzarotto C R, Malinin N L, Li Y, et al. CHANGE-seq reveals genetic and epigenetic effects on CRISPR-Cas9 genome-wide activity [J]. Nature Biotechnology, 2020, 38(11): 1317-1327. doi: 10.1038/s41587-020-0555-7 [41] Nidhi S, Anand U, Oleksak P, et al. Novel CRISPR-Cas systems: an updated review of the current achievements, applications, and future research perspectives [J]. International Journal of Molecular Sciences, 2021, 22(7): 3327. doi: 10.3390/ijms22073327 -





 下载:
下载: